量子力学/分子力学(QM/MM)方法是探索酶催化反应机理的重要计算工具,被广泛应用于药物设计和生物大分子反应研究。然而,近年来有研究指出,即使在相同体系和近似条件下,QM/MM计算结果也可能因软件实现差异而出现显著偏差,这给结果的可重复性与可靠性带来了挑战。
近日,吉林大学理论化学研究所的孙小丽副教授与瑞典隆德大学UIf Ryde教授合作在Journal of Chemical Theory and Computation发表的最新研究,对QM/MM方法在新冠病毒主蛋白酶(SARS-CoV-2 Mpro)体系中的可重复性进行了系统而深入的检验(图1)。
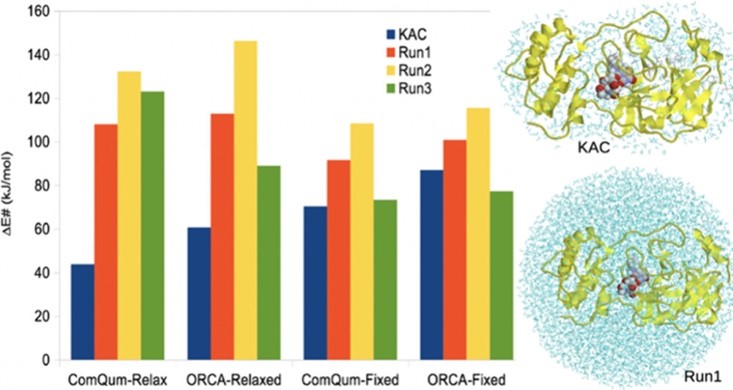
图1. 不同QM/MM软件与蛋白质模型下的活化能(ΔE‡)差异
研究选取NWChem、Q-Chem、ComQum、ORCA和AMBER五种主流软件,研究发现发现,不同软件在计算反应能(ΔE)与活化能(ΔE‡)的评估上存在显著差异。深入分析表明,这些差异主要源于电荷重分布方案(charge-redistribution scheme)的不同,同时加法/减法方案、嵌入方式(机械嵌入、静电嵌入、极化嵌入)及截断原子的处理方法也会对结果产生重要影响。因此明确QM/MM计算的细节至关重要。
此外,电荷重分布方案的巨大影响以及MM项的较大比重表明,所使用的83原子QM区域过小。为此,我们采用了大QM(big-QM)方法来获得对QM区域大小收敛的能量结果。在分析过程中,一个意外的不收敛现象揭示出裸露在溶剂中的带电残基对能量影响显著,而将其电荷中性化后,能量结果呈现出合理收敛趋势。我们建议在原有big-QM方案的基础上增加一个规则:在big-QM区域中,中和所有暴露于溶剂的带电残基。
最后,考察了不同蛋白质构建、溶剂化与优化策略(如水分子层数、晶体固定与放松)对QM/MM结果的影响更为突出,ΔE‡可相差88 kJ/mol,ΔE可相差114 kJ/mol。这表明,SARS-CoV-2主蛋白酶的反应位点位于蛋白表面,对溶剂结构极为敏感,仅基于最小化结构的静态计算难以捕捉真实反应路径,更适合采用自由能采样方法(如umbrella sampling或metadynamics)进行深入探索。
本研究成果为新冠病毒主蛋白酶抑制剂的设计及其他生物大分子反应机理研究提供了更加可靠的计算框架,对推动精准药物研发具有重要指导意义。
论文链接:https://pubs.acs.org/doi/10.1021/acs.jctc.5c00841